
 我要投稿
我要投稿
细胞生理、病理状态的改变是人体衰老、疾病发生发展的重要机制。
自1885年建立体外细胞培养技术以来,细胞作为模型系统已经广泛地用于疾病研究和药物筛选,是目前全球使用最广泛的体外研究模型。国际上,基于多种组学技术特别是基因表达谱技术的药物研究模式已成趋势。
基因表达是指储存遗传信息的基因经过一系列步骤表现出其生物功能的过程。特定基因表达量的多少,可以用于疾病的诊断、预后或预测治疗反应,在药物筛选、预测药物活性、发现药物新靶点等方面显示出了强大的生命力。
1665年,罗伯特·胡克在《显微术》一书中,首次描述并命名了细胞。1838年,Theodor Schwann和Matthias Schleiden提出了细胞学说的主要论点,指出细胞是生命结构和功能的基本单位,从而确定了细胞在生物学的核心地位。
随着现代细胞生物学和分子生物学的发展,细胞的结构和功能研究日趋详尽,越来越多的细胞类型得到鉴定,各种生命活动同细胞功能及细胞内部分子变化联系起来。2017年,美国启动“人类细胞图谱”计划,致力于绘制人体中的每种细胞类型,改变了我们对生物体、细胞和疾病的认识。
2022年5月,顶尖学术期刊Science四篇重磅论文同时上线,报告了构建综合性人类细胞图谱取得的里程碑结果,绘制出了迄今最为全面的“人类细胞图谱”,覆盖了100多万个细胞,包括来自68个捐赠者的33种组织,共计500多种人体细胞类型[1]。
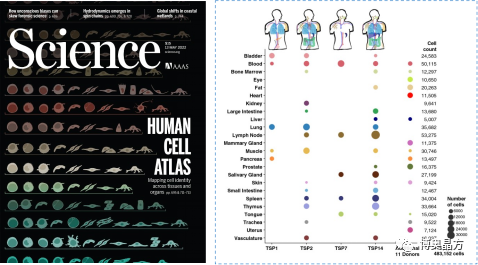
图1 2022年人类细胞图谱计划[1]
细胞通过对基因信息的表达调控,实现生长、代谢、分裂、分化、衰老、死亡等生理或病理过程,人体中不同的细胞组合在一起,相互作用,构成不同的器官,并执行不同的功能,从而决定着人体的生、老、病、死。
细胞功能的改变是驱动人体衰老和疾病发展的根本[2,3,4]。2023年1月3日Cell发表重磅综述文章《Hallmarks of aging: An expanding universe》,提出人体衰老的12个标志,主要涉及细胞层面或分子功能层面的异常改变[5]。
作为一切生命活动的基础,细胞是疾病发生的根源,疾病中的问题都要在细胞中去寻找致病的原因、发展的过程,以及治病的方法。
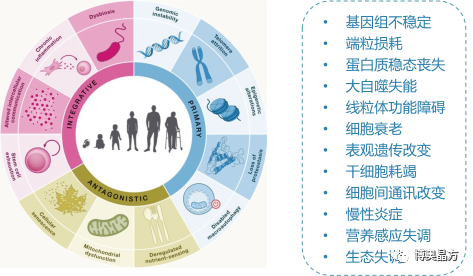
图2 细胞衰老是驱动人体各系统衰老的根本(衰老12大标志)[5]
药物筛选是药物发现的关键。理想的药物筛选技术不但需要有更高的筛选效率、更大的筛选规模和更快的筛选速度,还要其检测模型能够准确、全面地反映被筛药物的生物活性特征。
常规基于靶标活性或化合物与靶蛋白体外反应的筛选技术,虽然具有快速、微量的特点,但只能做到较少指标检测,提供化合物对靶点作用的有限信息,无法对化合物的生物活性进行综合评价。
动物模型一直是确定新药安全性和有效性的经典方法。但其效率低、成本高,也不太适合开展大规模、高通量的药物筛选。此外,动物与人类存在种属差异,动物模型在临床预测方面失败率较高。
细胞水平的活性筛选是连接分子靶标水平和动物(人体)水平活性的桥梁。自从1885年,德国学者Wilhelm Roux首次建立体外细胞培养技术以来,细胞作为模型系统已经广泛地用于疾病研究和药物筛选,已经成为目前全球使用最广泛的体外研究模型。
现代细胞培养技术的发展,细胞模型可以模拟人体各种器官、系统中的细胞变化,可以更好地模拟体内环境和药物代谢,从而更真实地反映药物在体内的效果。基于细胞开展药物筛选,可以比基于体外生化反应,更好地模拟体内环境和药物代谢,从而更真实地反映药物在体内的效果。
细胞易于培养,试验周期短,容易实现高度自动化、规模化,极大地提高了药物筛选效率和结果准确性。此外,细胞实验所需的样品极少(微克级),对某些昂贵或极难获取药物,具有重要价值。
细胞水平筛选体系常用技术包括高内涵显微成像、报告基因系统、荧光标记检测等,目前在靶向细胞活性、细胞因子、生长因子、离子通道等单一活性功能指标上,取得了较好的进展。
随着人类基因组计划的完成,基因组、转录组、蛋白组、代谢组等组学技术的发展,深刻揭秘了疾病和生命现象的根源,使得人们对细胞、基因与疾病的关系有了更深刻的理解。
组学技术在药物筛选中的应用,激发了许多新思路、新方法的探索和研究。组学技术可以相对快速地揭示药物作用机制,能够从多个角度、多个方面展现药物的作用效应,在药效评价、毒性筛选、分子机制研究以及药物代谢动力学等药物研究提供了全新的解决方案。将组学技术深入应用在药物筛选上,在筛选时间、精度、可控性和经济效益方面都具有明显的优势。
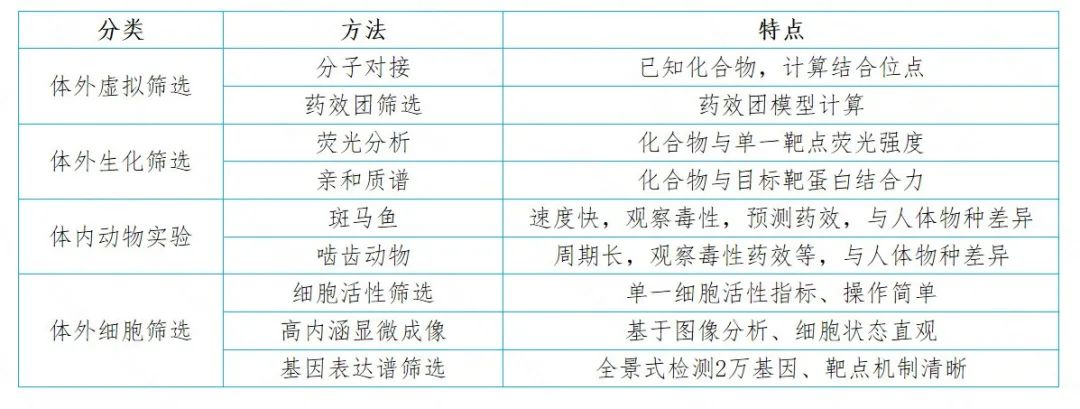
下表中对比了虚拟筛选、生化筛选、动物实验与细胞筛选的技术特点:
2006年,美国Broad研究所通过对1309种药物作用于5种人类肿瘤细胞系后的基因表达谱,建立了小分子药物处理人类细胞的“药物-疾病-基因”关联图谱CMap(Connectivity Map)[6]。这些数据库可以帮助研究人员快速发现与疾病有高关联性的小分子化合物或药物,或者预测出药物分子可能的作用机制,从而大幅推动药物研发的进展。
通过比较候选药物处理和疾病条件下细胞或组织的基因表达图谱,如果两者存在互补关系,则说明该候选药物可能对该疾病具有疗效;另一方面,候选药物的基因表达图谱可与CMap数据库中的药物或化合物的基因表达图谱做比对,进而确定候选药物的可能疗效与药理学机制。
基于CMap和基因集合富集分析方法,通过与不同细胞状态下的基因表达谱印记对比分析,成功发现了新的药物作用模式[7,8],在药物重定位[9]、疾病相关基因发现[10]、疾病机理研究[11]等方面得到了应用。
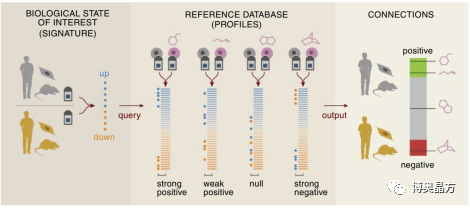
图3 CMAP原理示意图
此后,美国国立卫生研究院(NIH)启动了基于网络的细胞反应印记整合图书馆(Library of Integrated Network-based Cellular Signatures,LINCS)计划,旨在全面描述不同小分子化合物、配体以及基因沉默扰动下的多层次细胞反应(如转录物表达水平、蛋白质表达水平、细胞表型等)。LINCS采用了一种低成本基因表达谱测定技术(L1000技术),通过检测978个标志基因,外推出其他基因的表达量,从而使需要测量的基因数目大幅减小,从而达到控制成本的目的。
L1000数据库是CMAP数据库的扩展,主要是使用32855个小分子对99个细胞系进行处理,包含约1,000,000个表达谱[12]。LINCS计划所产生的细胞反应大数据已经显示出在基因调控关系挖掘、疾病发生和药物作用机理辨识、药物新用途发现等方面的广泛用途。
Young等[13]用LINCS中L1000里基因沉默扰动数据,推断基因间的调控关系,并且根据转录因子数据库TRANSFAC和JASPAR里所识别的关系得到验证。Xie等[14]利用L1000药物扰动下转录组水平的数据,系统发掘并预测了480种已上市药物用于其他治疗属性的潜力。Chen等[15]用L1000数据集系统研究并量化了药物与疾病扰动下基因表达特征之间的逆转关系,最终预测出4种化合物可有效逆转肝癌细胞中的基因表达,并在5种肝癌细胞系中得到验证。
中药作用机制研究一直是中医药现代化研究的关键问题之一。依据“君臣佐使”的原则,中药方剂常以多味药材组合入药,其作用机制复杂,使得中药研究面临较大的挑战。
2014年,程京院士提出建立分子版《本草纲目》研究计划(简称“分子本草”),该计划基于微量细胞的高通量药物转录组测序技术和人工智能技术,构建了具有自主知识产权的全转录组基因表达谱中药筛选平台。
该平台使用微孔板配合自动化工作站操作,仅需药物处理数千个人源细胞,经裂解后反转录、低循环PCR扩增、文库构建及测序过程,可进行全转录组的基因表达谱检测,为中药筛选和机制解析提供了一种全景式、高通量的药物筛选方法。
目前已构建了世界超大规模的中药分子功能基因表达谱数据库,该数据库真实检测了900多种中药、300多种食物提取物对人体9大系统疾病细胞模型作用的10亿级真实基因表达数据和2500多万条药用信号通路数据。
基于人工智能、高性能计算、分子组学图谱技术,结合中药知识图谱,以逆转疾病通路为核心,建立了人工智能驱动的中药创新研发平台,用数字化技术赋能中药创新药研发、药食同源健康食品开发、天然植物化妆品开发。
通过细胞基因表达谱技术,揭示药物与疾病的秘密,为中医药现代化和国际化开创全新的科学发展路径。
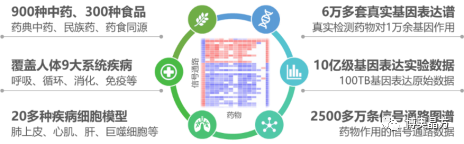
图4 分子本草平台世界超大规模中药药用基因表达谱数据库
已申请发明专利15项、获得软件著作权9项、发表SCI论文10 篇(总影响因子102.3)。获得中关村高新技术企业、双软认证企业、AAA级信用企业、博士后工作站资质。获得北京市知识产权局专利转化专项支持、入选中关村科技型小微企业关键技术创新支持项目,并获得科技部2022年全国颠覆性技术创新大赛总决赛最高奖,入选中华中医药学会2022年度“中医药十大学术进展”。
[1] Liu Z, Zhang Z. Mapping cell types across human tissues[J]. Science, 2022, 376(6594): 695-696.
[2] Zou Z, Long X, Zhao Q, et al. A single-cell transcriptomic atlas of human skin aging[J]. Developmental cell, 2021, 56(3): 383-397. e8.
[3] Abdellatif M, Rainer P P, Sedej S, et al. Hallmarks of cardiovascular ageing[J]. Nature Reviews Cardiology, 2023: 1-24.
[4] Wilson D M, Cookson M R, Van Den Bosch L, et al. Hallmarks of neurodegenerative diseases[J]. Cell, 2023, 186(4): 693-714.
[5] López-Otín C, Blasco MA, Partridge L, Serrano M, Kroemer G. Hallmarks of aging: An expanding universe. Cell. 2023 Jan 19;186(2):243-278.
[6] Lamb J, Crawford ED, Peck D, etal. The Connectivity Map: using gene-expression signatures to connect small molecules, genes, and disease. Science. 2006 Sep 29;313(5795):1929-35.
[7] Iorio F, Tagliaferri R, Di Bernardo D. Identifying network of drug mode of action by gene expression profiling. Journal of Computational Biology: A Journal of Computational Molecular Cell Biology, 2009, 16(2): 241-251.
[8] Iorio F, Bosotti R, Scacheri E, et al. Discovery of drug mode of action and drug repositioning from transcriptional responses. Proc Natl Acad Sci USA, 2010, 107(33): 14621-14626.
[9] Chang M, Smith S, Thorpe A, et al. Evaluation of phenoxybenzamine in the CFA model of pain following gene expression studies and connectivity mapping. Molecular Pain, 2010, 6(1): 56-69.
[10] Reka A K, Kuick R, Kurapati H, et al. Identifying inhibitors of epithelial-mesenchymal transition by connectivity map-based systems approach. Journal of Thoracic Oncology: Official Publication of the International Association for the Study of Lung Cancer, 2011, 6(11): 1784-1792.
[11] Wei G, Twomey D, Lamb J, et al. Gene expression-based chemical genomics identifies rapamycin as a modulator of MCL1 and glucocorticoid resistance. Cancer Cell, 2006, 10(4): 331-342.
[12] Subramanian A, Narayan R, Corsello S M, et al. A next generation connectivity map: L1000 platform and the first 1,000,000 profiles[J]. Cell, 2017, 171(6): 1437-1452.
[13] Young W C, Raftery A E, Yeung K Y. A Posterior probability approach for gene regulatory network inference in genetic perturbation data. Mathematical Biosciences & Engineering, 2017, 131(6): 1241-1251.
[14] Xie L, He S, Wen Y, et al. Discovery of novel therapeutic properties of drugs from transcriptional responses based on multi-label classification. Scientific Reports, 2017, 7(1): 7136-7147.
[15] Chen B, Ma L. Reversal of cancer gene expression correlates with drug efficacy and reveals therapeutic targets. Nature Communications, 2017, 8: 16022-16034.
